11月30日,国际学术期刊Nature Methods发表了中国科学院上海营养与健康研究院计算生物学研究所Andrew Teschendorff组的研究论文“Identification of differentially methylated cell types in epigenome-wide association studies”,报道了一种新的有助于识别与疾病相关的表观遗传通路的“CellDMC”统计算法,“破解”了表观全基因组关联分析中的关键难点。
DNA甲基化(DNAm)是DNA的共价修饰,其可以调节基因活性,并且易受到疾病风险因子的影响。表观全基因组关联分析(EWAS)的主要目标是测量大量个体中全基因组DNA甲基化,鉴定与疾病风险相关的DNA甲基化变化。然而,这一过程的主要障碍之一是细胞类型异质性:EWAS所使用的组织是不同细胞类型的复杂混合物,每种细胞类型具有其特有的DNA甲基化谱,因此可能产生错误的分析结论。到目前为止,尚无有效手段来确定驱使DNA甲基化发生变化的细胞类型和表观遗传通路。
在Andrew Teschendorff研究员的指导下,博士生郑世杰等人开发了一种名为“CellDMC”的新型统计算法,该算法不仅能够确定特定基因组位点的变化,而且能够确定导致这些DNA甲基化变化的细胞类型。相较于当前无法识别DNA甲基化变化的通用方法,这种算法对于DNA甲基化变化的识别灵敏度超过90% 。随后研究人员利用几个真实EWAS数据进行算法测试,结果表明类风湿性关节炎相关的大量DNA甲基化变化发生于一种特定的血细胞亚型(B细胞),这种B细胞的改变在该疾病的发生机制中特别重要。在另一个测试中,该算法识别了暴露于烟雾致癌物质的正常细胞和肺癌祖源细胞中的DNA甲基化变化,从而有助于将吸烟有关的表观遗传通路与肺癌进行关联。
值得一提的是,EWAS中有一些昂贵且难以实现的技术,如细胞分选技术,单细胞甲基化组测序技术。CellDMC可帮助EWAS的研究人员识别疾病相关细胞类型的改变,而无需使用这些技术。检测疾病相关和疾病风险相关的细胞类型改变的能力,对于鉴定和开发表观遗传疾病风险生物标志物,以及实现P4医学目标具有重要意义。
上述研究得到了国家自然科学基金委员会(31571359;31771464; 31401120)和中国科学院的资助。
论文链接:https://www.nature.com/articles/s41592-018-0213-x
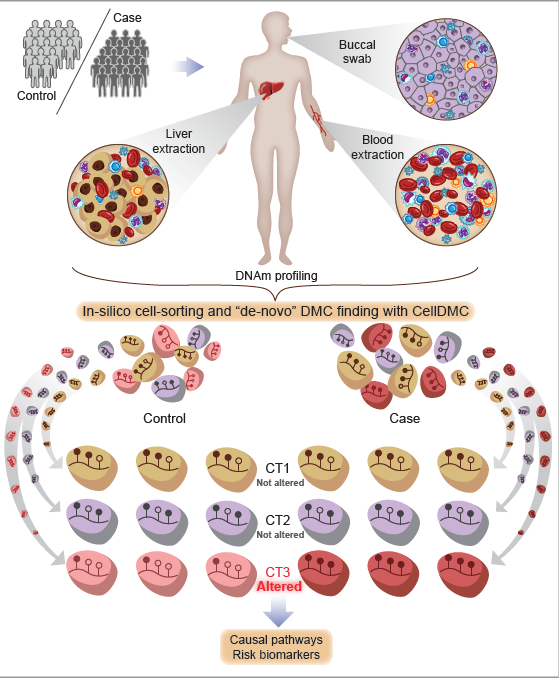
图. CellDMC 识别差异化甲基化的细胞类型