2022年2月28日,Genome Biology在线发表了中国科学院上海营养与健康研究所邵振研究组题为“HyperChIP: identification of hypervariable signals across ChIP-seq or ATAC-seq samples”的方法学论文,报道了其开发的HyperChIP计算模型。它能对多个ChIP/ATAC-seq样本间信号强度差异进行统计建模,识别高变信号基因组区域(hypervariable regions,HVRs);一系列下游分析表明,这些表观调控信号样本间差异显著高于背景模型的位点可有效用于揭示被比较样本集的宏观异质性结构(疾病亚型或进程、组织分化阶段、种群遗传背景等)及其上游调控机制。
真实人群尤其是癌症等慢性疾病病人在基因组、转录组等很多层面表现出较高的异质性。它们紧密联系着疾病临床表现等个体表型差异。但由于表观组ChIP-seq和ATAC-seq数据跨样本定量比较还存在许多公认难题,尚未有能通过多样本统计比较来识别高异质性表观调控位点的完整计算模型被发表,造成很多相关研究只能采用一些经验方法。但是,这些经验分析方法还存在易受技术因素影响和缺乏统计显著性判据等缺陷,成为制约疾病和正常人群表观组异质性精准解读的一个关键技术瓶颈。
针对这一点,HyperChIP提出使用拟合的ChIP/ATAC-seq信号强度均值-方差关系来对每个位点的观测信号方差进行标度,并以基因表达数据为参照,指出相比于各种经验统计指标,上述方法获得的标度方差(scaled variance)能更好地衡量每个位点表观调控信号的样本间异质性水平(图A) ;然后,在评估其统计显著性时,引入区域筛选(subset selection)和缩尾处理(winsorization)等手段来控制潜藏真阳性位点对参数估计的影响,显著改善了HVR识别的统计功效,实现对被比较样本间表观组异质性的完整统计刻画(图B)。
将HyperChIP应用于42例肺腺癌病人肿瘤的H3K27ac ChIP-seq数据,发现在识别的HVRs中,很多位点的H3K27ac水平与肿瘤的临床诊断阶段有较强的关联。另一方面,应用它分析由数百例TCGA肿瘤样本生成的大型pan-cancer ATAC-seq数据集,发现基于所得HVRs能较好地将不同癌症类型的样本分开。其中一些组织来源或细胞形态相近而聚在一起的癌症样本,被归类定义为四个癌症超类型(super class),即脑部肿瘤、消化道腺癌、肾癌和鳞状细胞癌。进一步,通过构建转录因子活性(TF activity)打分模型,发现了大量被特定癌症超家族中样本所共有的转录调控因子(图C)。它们中很多被基因表达数据和其它研究结果支持。例如,TP63被发现在鳞细胞癌样本间具有普遍较高的转录因子活性,而很多研究已经指出它是一个泛鳞癌致癌因子。
此外,将HyperChIP应用于正常组织ChIP/ATAC-seq数据也能获得有价值的发现。例如,应用它比较着床前不同时间点的小鼠胚胎ATAC-seq数据,对所得的HVRs进行主成分分析,发现第一主成分指征了胚胎的不同发育阶段;进一步结合转录因子活性分析,可有效发掘出不同阶段所特异的转录调控因子。另一方面,应用它比较十余个不同人类个体的淋巴母细胞系CTCF ChIP-seq数据,对所得的HVRs进行主成分分析,发现能很好地将样本按个体所属种群分开(图D)。
中国科学院上海营养与健康研究所博士生陈浩杰和博士后涂世奇为该论文共同第一作者,邵振研究员和涂世奇为共同通讯作者。复旦大学生命科学学院张一婧研究员、复旦大学附属肿瘤医院孙艺华主任等对该工作的提出和完善也做出了重要贡献。该工作获得了国家自然科学基金委,科技部,中国科学院的资助。上述研究所使用的所有数据均来自已公开发表数据。
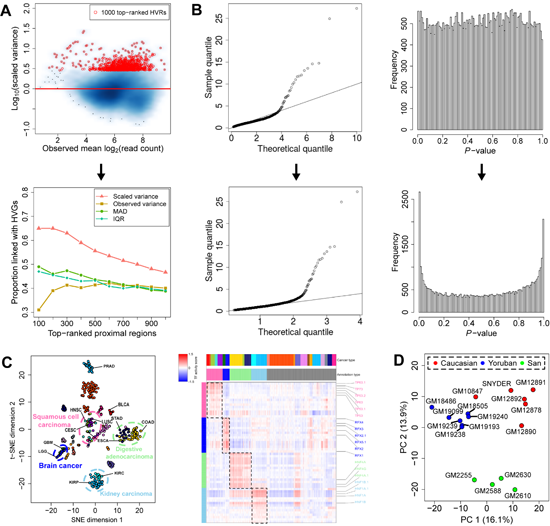
图:(A) HyperChIP模型以校正了均值-方差关系的标度方差(scaled variance)为统计指标, 使不同信号强度水平的基因组区域之间的信号差异水平更加可比。(B)通过引入区域筛选和缩尾处理等手段来控制潜藏真阳性对参数估计的影响,显著改善了HVR识别的统计功效。(C)将HyperChIP应用于TCGA pan-cancer ATAC-seq数据集,基于所得HVRs将部分组织来源或细胞形态相近的样本归类定义为四个癌症超类型(super class),即脑部肿瘤(Brain cancer)、肾癌(Kidney carcinoma)、消化道腺癌(Digestive adenocarcinoma)和鳞状细胞癌(Squamous cell carcinoma);下游的转录因子活性打分分析,发现了大量被每个超家族中肿瘤样本所共有的转录调控因子。(D)比较十余个不同人类个体的淋巴母细胞系CTCF ChIP-seq数据,发现基于所得HVRs能很好地区分不同种群个体的样本。
论文链接:https://genomebiology.biomedcentral.com/articles/10.1186/s13059-022-02627-9