2月9日和12日,中科院上海营养与健康研究所杨力研究组受邀在SCIENCE CHINA Life Sciences和Methods杂志分别发表题为“Fast and Furious: insights of back splicing regulation during nascent RNA synthesis”的综述论文和“CIRCexplorer pipelines for circRNA annotation and quantification from non-polyadenylated RNA-seq datasets”的方法论著文章。
已知主要有两类RNA剪接相关的环形RNA,包括外显子反向剪接形成的环形RNA(circRNA)和内含子剪接后加工成熟的环形RNA(ciRNA),其以特征的共价闭合环形结构存在,不具有经典的5'帽子和3'尾巴。大数据分析在体内存在数以万计的circRNA,解析这些circRNA的生成加工及功能作用是领域的研究前沿热点。针对circRNA产生效率低的现象,第一篇综述论文主要从新生circRNA加工的角度阐述了转录过程中反向剪接催化调控的机制(图示A),通过对不同新生RNA-seq测序技术特点的分析指出4sUDRB-seq测序方法可以更好地用于新生circRNA的调控研究。最后,该综述论文也对利用4sUDRB-seq开展另一种特殊形式的剪接事件-递归剪接(recursive splicing)的分析进行了介绍和讨论。第二篇方法论著文章主要集中对circRNA计算分析方法及其具体流程进行介绍。针对可用于环形RNA分子(包括circRNA和ciRNA)发掘的多种类型高通量转录组测序数据(包括poly(A)– RNA-seq, ribo– RNA-seq和RNase R RNA-seq等),研究人员选取了研究组前期发展的一系列高效计算分析流程CIRCexplorer(version 1-3)进行详细的计算分析流程展示,对其在circRNA分析中的应用场景进行阐述,包括环形RNA的注释,可变(反向)剪接事件的鉴定和环形RNA与其对应线形RNA的定量比较等(图示B),为开展circRNA的计算分析提供了清晰明了的操作指南。
中科院上海营养与健康研究所杨力组博士生薛尉和博士后马旭凯分别为上述两篇文章的第一作者,杨力研究员为通讯作者。此外,杨力组硕博研究生南芳作为共同第一作者参与撰写了circRNA结构解析的方法论著文章发表在2月9日的Methods杂志上。杨力组的相关工作受到国家自然科学基金委、科技部和中科院青促会等项目的支持。
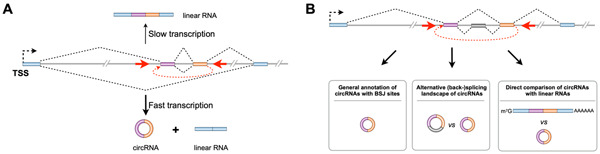
图示:(A) RNA Pol II的快速转录会促进环形RNA的生成。(B) 环形RNA注释、可变反向剪接事件和环形RNA与其对应线形RNA精准定量及比较。
原文链接:
Xue et al., Sci China Life Sci 2021
http://engine.scichina.com/doi/10.1007/s11427-020-1881-1
Ma et al., Methods 2021
https://www.sciencedirect.com/science/article/pii/S1046202321000451
Guo et al., Methods 2021
https://www.sciencedirect.com/science/article/pii/S1046202321000232